前面已经分享过对多模态MR图像脑肿瘤进行分割处理的例子。今天将继续分享使用多分类Focalloss函数来训练分割网络。为了方便大家学习理解整个分割流程,我将整个流程步骤进行了整理,并给出每个步骤的结果,希望对大家有所帮助。
一、脑肿瘤图像分析与预处理
(1)、多模态MR脑肿瘤图像分析。
分析的过程基本上跟上一篇一致,这里就不多言了,直接从数据处理开始。
(2)、准备脑肿瘤分割数据。
首先将4个模态序列的MR原始图像进行合并生成4个通道的三维图像,原始图像大小都是(240x240x155x1),合并后大小是(240x240x155x4);
其次对Mask图像进行one-hot操作,将原始图像大小都是(240x240x155x1),生成大小是(240x240x155x4):通道0中非零值区域是背景区域,通道1中非零值是坏疽区域,通道2中非零值是浮肿区域,通道3中非零值是增强肿瘤区域;
最后对图像和Mask进行分块——取Patch操作,生成若干个(128,128,64)大小的图像和Mask,判断并输出非零的Mask和对应的图像。
二、脑肿瘤分割
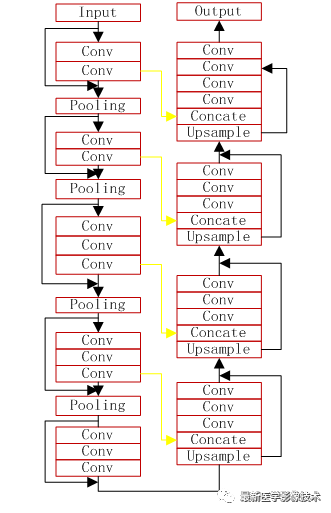
(1)、搭建VNet3d模型,与二分类VNet3d模型的不同之处就在于最后一层输出采用SoftMax激活函数,网络输入大小是(128x128x64),loss采用的是多类Focalloss函数。
多类Focalloss函数tensorflow版本实现如下所示:
def categorical_focal_loss(Y_pred, Y_gt, gamma, alpha):
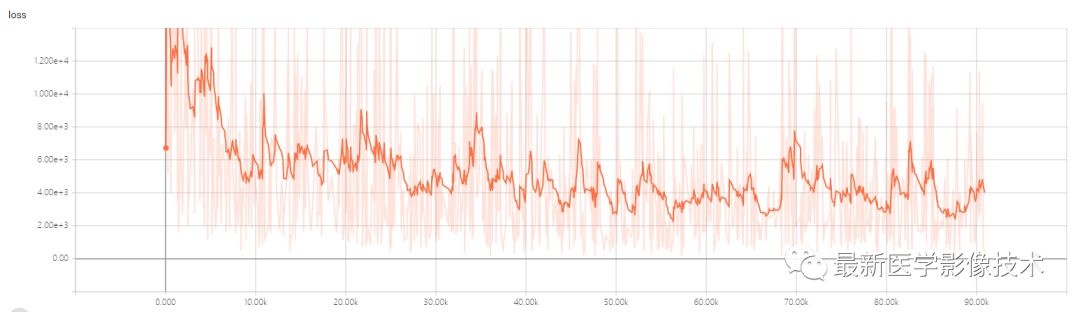
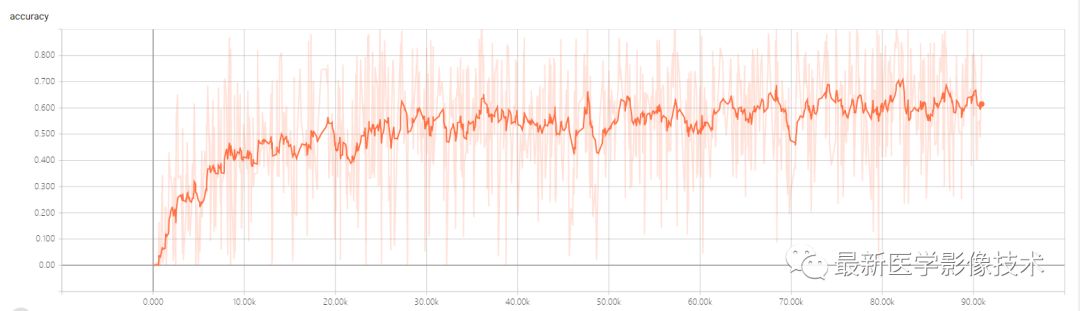
(2)、训练的损失函数和精度如下图所示。
(3)、脑肿瘤分割推理过程:首先将原始flair,T1,T2,T1ce图像一起读取进来并进行z-score标准化操作,然后将四个模态图像合并成4通道三维图像(240x240x155x4),输入到网络中去,网络输入大小是(240x240x48x4),在z方向上分块输入并拼接最终得到(240x240x155)分割结果。
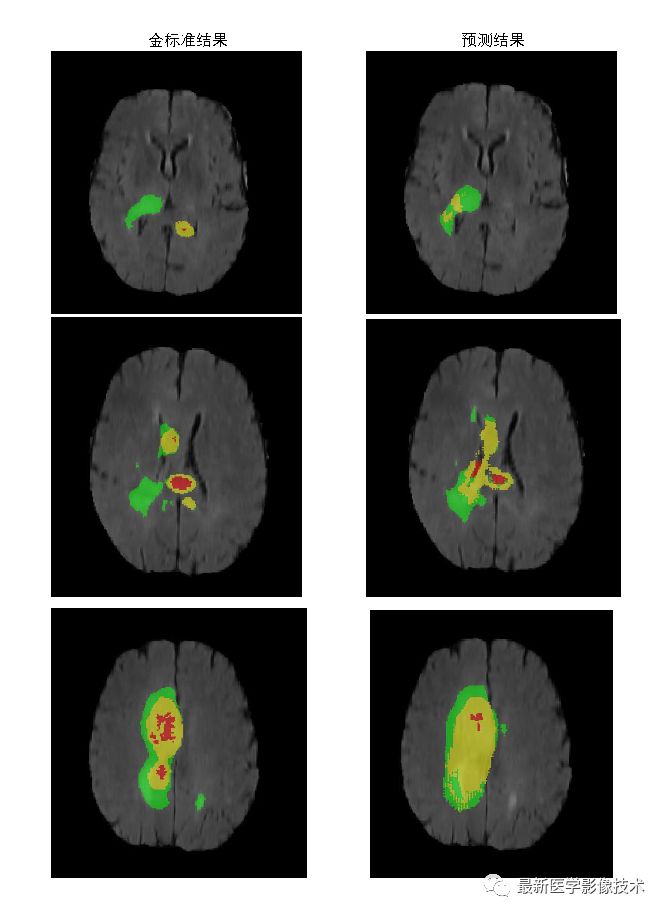
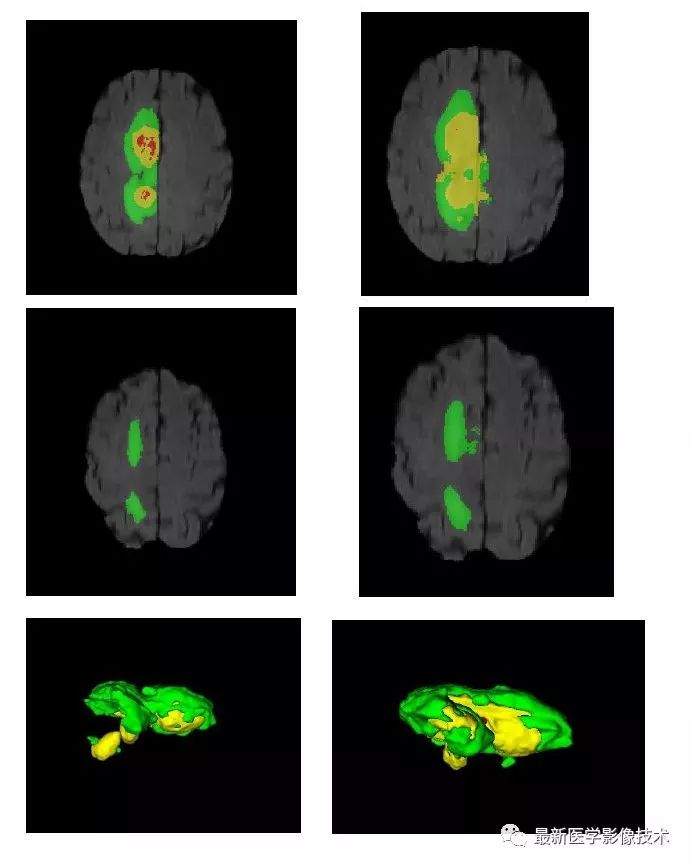
(4)、进行了结果测试,左边是金标准图像,右边是预测结果图像,如下所示。
为了大家更好的学习,我把整个项目代码分享到github上:
https://github.com/junqiangchen/BraTS18-Challege
如果大家觉得这个项目还不错,希望大家给个Star并Fork,可以让更多的人学习。如果碰到任何问题,随时留言,我会尽量去回答的。
本文分享自微信公众号 - 最新医学影像技术(MedicalHealthNews)。
如有侵权,请联系 support@oschina.cn 删除。
本文参与“OSC源创计划”,欢迎正在阅读的你也加入,一起分享。